Laissons la parole aux chercheurs, voici un article publié par Le Journal du CNRS sous la plume de Jonathan Rangapanaiken.
En une semaine seulement, des scientifiques français ont mis en place un projet pluridisciplinaire de criblage virtuel à grande échelle afin d’identifier, d’ici à dix-huit mois, des candidats susceptibles d’inhiber le virus SARS CoV-2 parmi 1,5 milliard de petites molécules.
Afin de trouver de nouvelles molécules ayant une activité sur le virus SARS-CoV-2 (i-e le Coronavirus CoVID19, Ndlr), un réseau de biophysiciens, biologistes, informaticiens et médecins, s’est structuré en seulement une semaine. Leur but : cribler virtuellement 1,5 milliard de molécules, c’est-à-dire les tester à l’aide de l’intelligence artificielle. « L’originalité de notre approche “virtuelle” vis-à-vis d’autres initiatives, c’est de coupler une échelle ultra-large de criblage avec les connaissances du virus de biologistes et de médecins. Car ensuite, les candidats potentiels seront synthétisés, purifiés et testés “en vrai” », explique Jean-Hugues Renault, spécialiste de chimie des substances naturelles à l’Institut de chimie moléculaire de Reims1 (ICMR), et porteur du projet.
Disposer de tels moyens ne serait pas possible en temps normal. Ce qui est absolument remarquable, c’est d’observer qu’en cas d’urgence, en très très peu de temps, une mobilisation et une réactivité exceptionnelles sont possibles.
Tout commence par quelques appels téléphoniques. Après des échanges et des courriels jusqu’au milieu de la nuit, Jean-Hugues Renault, qui est aussi directeur de l’ICMR, monte un projet de criblage virtuel ultra-large en sept jours et le dépose auprès de l’Agence nationale de la recherche… dix minutes avant la fermeture de l’appel !
Pour lui, c’est du jamais vu : en un temps record, le projet appelé HT-Covid2 a mobilisé six laboratoires de recherche3, un CHU4 et plusieurs grandes infrastructures de recherche5 parmi lesquelles la plateforme ChemBioFrance du CNRS, dédiée au criblage haut-débit. Et surtout, trois centres de calcul français6 qui mettent à disposition jusqu’à 115 000 processeurs et 1,5 pétaoctet (soit 1,5 million de gigaoctets) de stockage.
Pour la plupart des protagonistes, c’est la première fois qu’ils travaillent ensemble. Le CNRS finance une partie du projet ainsi que la région Grand Est. Et à l’heure actuelle, plusieurs centaines de millions de molécules ont déjà été criblées.
Deux stratégies de criblage
Et l’enjeu est majeur dans la lutte contre la pandémie. « Pour lutter contre le SARS-Cov-2, il y a trois possibilités : trouver un vaccin, piocher dans la pharmacopée existante ou trouver de nouvelles molécules ayant une activité antivirale. C’est dans ce troisième cas que notre criblage virtuel ultra-large entre en jeu », explique Jean-Hugues Renault.
Mais le criblage virtuel, c’est quoi ? « À l’aide de moyens informatiques assistés par l’intelligence artificielle, on teste un très grand nombre de molécules naturelles ou synthétiques. On identifie si elles possèdent une activité biologique intéressante, par exemple pour inhiber la réplication de SARS CoV-2. Il est clair qu’à ce stade, l’apport des collègues biologistes et médecins est essentiel », explique le chercheur.
Une grande puissance de calcul va permettre de simuler à grande vitesse l’activité que pourrait avoir une molécule sur l’une ou l’autre des protéines d’un virus. Un peu comme si on sous-traitait à 115 000 processeurs une partie du processus long et coûteux d’identification de médicaments candidats.
Seconde salle informatique du centre de calcul de l’IN2P3. Ce centre, ainsi que deux autres, mettent leurs processeurs à disposition du projet HT-Covid pour simuler à grande vitesse l’activité des molécules candidates sur les protéines du virus.
C. Fresillon/CC IN2P3 Photothèque
En plus de la puissance exceptionnelle de calcul mobilisée, l’originalité du projet HT-Covid réside dans une double approche du criblage virtuel permettant d’explorer un « espace chimique » extrêmement large.
D’un côté, 70 000 composés existants et répertoriés dans des bases de données ou des collections comme celle de la Chimiothèque nationale, sont testés in silico (c’est-à-dire par ordinateur) sur plusieurs cibles protéiques connues et pertinentes du virus. « Chaque semaine, de nouvelles données sur la structure du virus sont publiées tant la recherche se mobilise sur la question », se réjouit Jean-Hugues Renault.
En parallèle, un criblage virtuel ultra-large est mené sur une seule cible, l’une des plus intéressantes du virus : 1,5 milliard de structures moléculaires, conçues par intelligence artificielle et pouvant être synthétisées rapidement par des robots, sont testées sur l’ARN réplicase à l’origine de la réplication du virus.
Une étude croisée des résultats des deux approches permettrait d’obtenir une sélection de cinq cents candidats pour la prochaine étape : le passage vers le monde réel. À savoir la fabrication de ces molécules et leur évaluation biologique.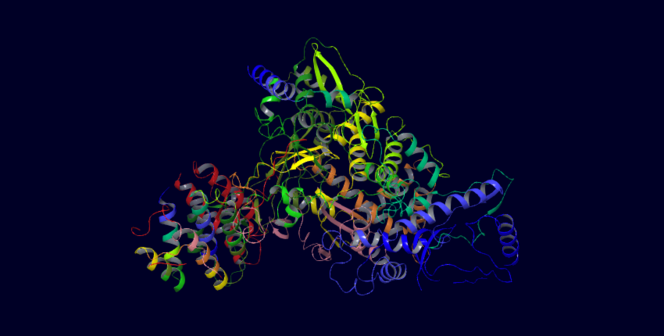
Cette ARN réplicase, ou ARN polymérase ARN-dépendante, est à l’origine de la réplication du virus. 1,5 milliard de structures moléculaires, conçues par intelligence artificielle, seront testées virtuellement sur elle. ©JH Renault Partager
Une mobilisation sans précédent
Ainsi à l’issue du criblage, certaines structures moléculaires identifiées peuvent déjà exister dans le commerce ou les chimiothèques. Sinon il faudra les synthétiser, les purifier avant de les soumettre aux étapes d’essais in vitro et in vivo. Pour Jean-Hugues Renault, « il n’y aura pas nécessairement de miracle en dix-huit mois, il s’agit d’une approche interdisciplinaire qui comporte des aspects de recherche fondamentale ».
Mais cette nouvelle méthodologie vient en parfait complément aux démarches académiques ou industrielles qui sont menées, et surtout, elle pourrait être transposée à l’identique pour d’autres pandémies. « Disposer de tels moyens ne serait pas possible en temps normal. Ce qui est absolument remarquable, c’est d’observer qu’en cas d’urgence, en très très peu de temps, une mobilisation et une réactivité exceptionnelles sont possibles. Les accords du Genci, du Centre de calcul de l’IN2P3 et du supercalculateur Romeo pour mettre leur puissance de calcul à disposition ont été immédiats et décisifs. » Chacun de ces centres a mis temporairement de côté certaines de ses expériences à la faveur de la lutte contre le Covid-19.
Un autre aspect important du projet : l’open data. Les données produites par le projet HT-Covid pourront être en accès libre. Une manière aussi d’affirmer que la sortie de cette crise devra être une opportunité pour faire évoluer les pratiques, même au sein de la recherche.
♦Notes
1.Unité CNRS/Univ. de Reims Champagne-Ardenne.
2.Le projet HT-Covid a été retenu sur la liste complémentaire de l’appel Flash Covid-19 de l’Agence nationale de la Recherche 2020.
3.IRCM ; Laboratoire d’innovation thérapeutique (LIT – CNRS/Univ. de Strasbourg) ; Chimie et interdisciplinarité : synthèse, analyse, modélisation (Ceisam – CNRS/Univ. de Nantes) ; Matrice extracellulaire et dynamique cellulaire (MEDyC – CNRS/Univ. de Reims Champagne-Ardenne) ; Infections cardiovasculaires virales et inflammation en pathologie humaine (CardioVir – Univ. de Reims Champagne-Ardenne) ; Centre de recherche en science et technologie de l’information et de la communication (Crestic – Univ. de Reims Champagne-Ardenne).
4.Service des maladies infectieuses du CHU de Reims.
5.ChemBioFrance via la plateforme Antiviral Drug Design Plateform du laboratoire Architecture et fonction des macromolécules biologiques (CNRS/Aix-Marseille Université) et la Chimiothèque nationale du CNRS ; plateforme Chem-Symbiose (CNRS/Univ. de Nantes).
6.Centre de calcul de l’Institut national de physique nucléaire et de physique des particules (IN2P3) du CNRS ; le Grand équipement national de calcul intensif (Genci), via le Centre informatique national de l’enseignement supérieur (Cines) à Montpellier ; Centre de calcul régional Romeo (près de Reims).


